Reakcja Łańcuchowa Polimerazy (ang. Polymerase Chain Reaction, PCR) to jedna z najczęściej wykorzystywanych technik biologii molekularnej.Pozwala ona na szybkie powielenie (amplifikację) specyficznego fragmentu DNA in vitro. Opracowana w 1983 roku przez Kary’ego Mullisa, stała się kamieniem węgielnym współczesnej diagnostyki, badań naukowych i kryminalistyki. Patrząc na gamę zastosowań, powszechność nie dziwi, że Mullis otrzymał za to odkrycie nagrodę Nobla.
Jak działa PCR?
Podstawą techniki PCR jest reakcja chemiczna katalizowana przez polimerazę łańcuchową. Polega ona na przyłączaniu komplementarnych nukleotydów do jednoniciowego DNA. Kluczem do wykorzystania tej reakcji w warunkach in vitro jest wykorzystanie enzymu, który jest odporny na działanie wysokiej temperatury. Pochodzi on z bakterii Thermus aquaticus występujących w gorących źródłach. Dzięki temu możliwe jest przeprowadzenie denaturacji dwuniciowego DNA niezbędnego by rozpocząć reakcję. Denaturacja zachodzi w temperaturze powyżej 90 stopni.
PCR opiera się na cyklicznym powtarzaniu trzech głównych etapów, które zachodzą w programowalnym urządzeniu zwanym termocyklerem.
Reakcja wymaga obecności następujących składników:
- Matrycowy DNA: Fragment DNA, który ma zostać powielony.
- Startery (primery): Dwie krótkie, jednoniciowe sekwencje DNA (ok. 18-30 nukleotydów), komplementarne do przeciwległych końców amplifikowanego fragmentu. Wyznaczają one początek syntezy.
- Nukleotydy (dNTP): Związki budulcowe (A, T, C, G) do syntezy nowej nici DNA.
- Termostabilna polimeraza DNA (np. Taq polimeraza): Enzym katalizujący syntezę nowej nici DNA, odporny na wysokie temperatury denaturacji.
- Bufor: Zapewnia optymalne środowisko (pH, stężenie soli) dla enzymu.
- Jony magnezu Mg2+: Kofaktor niezbędny do prawidłowej pracy polimerazy.
Etapy reakcji PCR
- Denaturacja (ok. 92 °C – 98 °C): W tej temperaturze dochodzi do zerwania wiązań wodorowych stabilizujących podwójną helisę DNA. Dwuniciowe DNA rozplata się na dwie nici.
- Hybrydyzacja (Annealing) (50 °C – 65 °C): Temperatura zostaje obniżona, umożliwiając starterom (primerom) przyłączenie się (hybrydyzację) do komplementarnych sekwencji na jednoniciowej matrycy.
- Elongacja (Wydłużanie) (ok. 72 °C): Polimeraza DNA dobudowuje nowe nukleotydy, rozpoczynając od końca 3′ startera, syntetyzując nową, komplementarną nić DNA.
Każdy cykl podwaja ilość amplifikowanego DNA, prowadząc do wykładniczego wzrostu liczby kopii. Proces jest powtarzany zazwyczaj 20-40 razy.
Jak zaprojektować startery?
Prawidłowe zaprojektowanie starterów jest kluczowe dla specyficzności i wydajności reakcji PCR. Główne zasady to:
- Długość: Startery powinny składać się z 18-30 nukleotydów.
- Temperatura topnienia (Tm): Powinna być zbliżona dla obu starterów (np. w zakresie 55 ° – 65 °C). Różnica nie powinna przekraczać 5 °C.
- Zawartość GC: Optymalnie par GC w starterach to 40-60%. Zbyt duża zawartość G/C może prowadzić do silniejszej hybrydyzacji.
- Unikanie struktur drugorzędowych: Startery nie powinny tworzyć między sobą struktur (np. dimerów starter-starter) ani w obrębie własnej cząsteczki (np. spinki do włosów), zwłaszcza na końcu 3′. Dimerizację starterów na końcu 3′ jest szczególnie krytyczna, ponieważ polimeraza może zainicjować syntezę od takiego miejsca, tworząc niepożądane produkty.
- Specyficzność: Startery muszą być komplementarne wyłącznie do docelowej sekwencji matrycowego DNA.
Jak uzyskać wynik?
Sposób analizy wyników zależy od rodzaju przeprowadzonej reakcji PCR:
- Konwencjonalny PCR (End-point): W klasycznej reakcji PCR należy wykonać jeszcze analizę próbki by uzyskać wynik. Najczęściej produkty reakcji są wizualizuje sięi rozdziela metodą elektroforezy w żelu agarozowym lub poliakrylamidowym. Fragmenty DNA migrują w żelu pod wpływem pola elektrycznego, a ich masa molekularna jest określana w stosunku do wzorca (drabinki mas). Uwidocznienie prążków (produktów amplifikacji) następuje po barwieniu DNA (np. bromkiem etydyny) i naświetlaniu światłem UV.
- qPCR (Real-Time PCR): Analiza opiera się na wspomnianej wcześniej wartości Ct oraz na krzywych topnienia (Tm) dla barwników niespecyficznie wiążących DNA, co pozwala na ocenę czystości i specyficzności produktu.
- dPCR (digital PCR): Wynik jest analizowany statystycznie na podstawie liczby pozytywnych mikroreakcji, dostarczając bezwzględną liczbę kopii DNA w próbce.
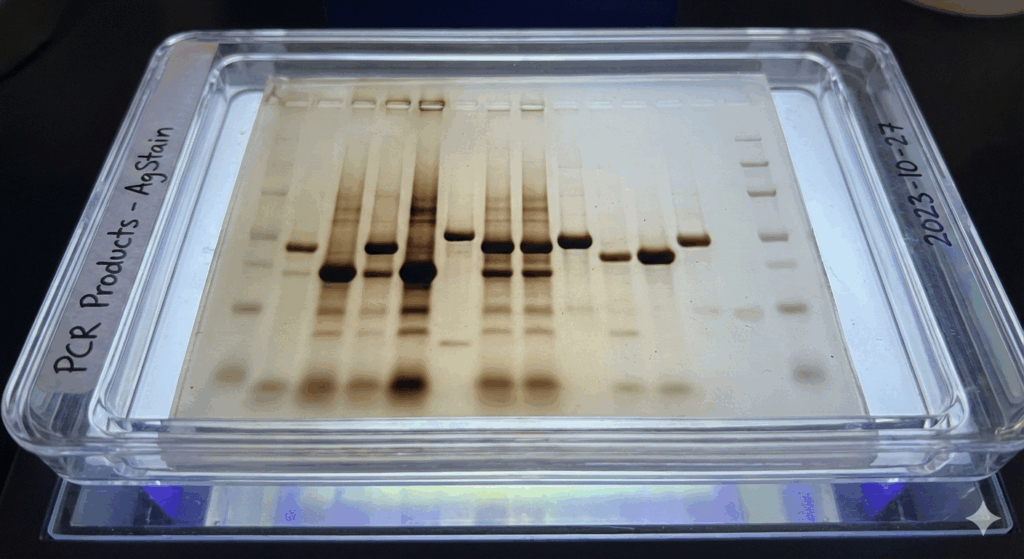
Modyfikacje PCR
Technika PCR ewoluowała, dając początek wielu modyfikacjom, które zwiększają jej zastosowanie i precyzję.
RT-PCR (Reverse Transcription PCR)
Polega na przepisaniu RNA na cDNA (komplementarny DNA) za pomocą enzymu odwrotnej transkryptazy (Reverse Transcriptase), a następnie przeprowadzeniu standardowej reakcji PCR.
Umożliwia to analizę ekspresji genów (mierzenie poziomu mRNA) oraz wykrywanie genomów wirusów RNA (np. SARS-CoV-2). Jest to kluczowa technika w diagnostyce wirusologicznej.
qPCR (Real-Time PCR)
qPCR nazywany jest również Real-Time PCR lub PCR w czasie rzeczywistym. W przeciwieństwie do tradycyjnego PCR, umożliwia ilościową analizę amplifikowanego produktu w trakcie trwania reakcji, a nie dopiero po jej zakończeniu. W tym celu wykorzystuje barwniki fluorescencyjne (np. SYBR Green) lub specjalne sondy (np. TaqMan), których fluorescencja jest monitorowana w każdym cyklu. Im więcej zamplifikowanego materiału tym silniejszy jest poziom fluorescencji.
Wynik ilościowy opiera się na cyklu progowym (Ct), czyli cyklu, w którym fluorescencja przekracza poziom tła. Im mniejsza wartość Ct, tym więcej początkowej matrycy było w próbce.
dPCR (digital PCR)
dPCR to najbardziej czuła metoda ilościowego oznaczenia DNA. Polega na rozdzieleniu mieszaniny reakcyjnej na tysiące (lub miliony) mikroskopijnych, oddzielnych reakcji (kropli lub studzienek). W każdej mikroreakcji następuje amplifikacja, a na koniec zlicza się te, w których nastąpiła amplifikacja (wynik pozytywny, „1”) i te, w których nie (wynik negatywny, „0”). Pozwala na bezpośrednie zliczenie absolutnej liczby cząsteczek matrycy DNA bez konieczności użycia krzywej standardowej, co czyni ją niezwykle precyzyjną.
Inne Modyfikacje
Multipleks PCR: Umożliwia amplifikację wielu różnych fragmentów DNA w jednej probówce, używając wielu par starterów.
Nested PCR (Zagnieżdżony PCR): Wykorzystuje dwie pary starterów i dwie kolejne rundy amplifikacji, co zwiększa specyficzność i czułość reakcji, redukując niespecyficzne produkty.
Zastosowanie PCR
Technika PCR zrewolucjonizowała wiele dziedzin nauki i praktyki.
Badania Naukowe
- Klonowanie i inżynieria genetyczna: Namnażanie konkretnych genów do dalszych manipulacji, modyfikacji i wklonowania do wektorów ekspresyjnych.
- Analiza ekspresji genów: Wykorzystanie RT-qPCR do ilościowego pomiaru poziomu transkrypcji genów (mRNA).
- Badania filogenetyczne i ewolucyjne: Analiza DNA w celu określenia pokrewieństwa między gatunkami.
- Mutageneza: Wprowadzanie specyficznych zmian w sekwencji DNA.
Kryminalistyka
- Identyfikacja osobnicza: Analiza krótkich powtórzeń tandemowych (STR) z minimalnych próbek materiału biologicznego (np. śliny, krwi, włosów) w celu stworzenia profilu genetycznego (DNA fingerprinting) dla identyfikacji sprawców lub ofiar przestępstw.
- Ustalanie ojcostwa: Porównanie profili genetycznych.
Farmacja i Medycyna
- Diagnostyka chorób zakaźnych: Szybkie i czułe wykrywanie materiału genetycznego patogenów (wirusów, bakterii, grzybów, np. HIV, SARS-CoV-2, borelioza) w próbkach klinicznych.
- Diagnostyka onkologiczna: Wykrywanie mutacji i zmian genetycznych związanych z nowotworami.
- Farmakogenomika: Identyfikacja wariantów genów wpływających na odpowiedź pacjenta na leki.
- Monitorowanie przeszczepów: Wykrywanie i pomiar ilości materiału genetycznego dawcy w celu wczesnego wykrycia odrzucenia przeszczepu.
Inne Zastosowania
- Rolnictwo i weterynaria: Identyfikacja odmian roślin, patogenów i zwierząt.
- Archeologia: Analiza starożytnego DNA (aDNA).
- Kontrola jakości żywności: Wykrywanie zanieczyszczeń mikrobiologicznych lub fałszerstw (np. identyfikacja gatunku mięsa).
Źródła
- Mullis K. B. (1990). The unusual origin of the polymerase chain reaction. Scientific American, 262(4), 56-65.
- Innis M. A. et al. (1990). PCR Protocols: A Guide to Methods and Applications. Academic Press. (Książka)
- Kubista M. et al. (2006). The real-time polymerase chain reaction. Molecular Aspects of Medicine, 27(2-3), 95-125.
- https://www.sigmaaldrich.com/PL/pl/technical-documents/protocol/genomics/pcr/standard-pcr
- https://www.sigmaaldrich.com/PL/pl/technical-documents/protocol/genomics/pcr/pcr-qpcr-dpcr-assay-design
- Taylor S. D. et al. (2019). Digital PCR: a critical review of theory and application. Biomolecular Detection and Quantitation, 18, 100080.
- https://zpe.gov.pl/pdf/P1HrrXrst
- https://www.rcsb.org/structure/1TAQ
